ポリマーナノ構造上のU2OS細胞におけるアクチンと接着斑組織の分析
要約
背景
この作業では、U2OSセルが平らなガラス表面に製造されたポリマーナノピラーのアレイによってどのように影響を受けるかを探ります。アクチン細胞骨格の組織と接着斑の位置、数、形状の変化を説明することに焦点を当てています。私たちの調査結果から、ナノピラー上での拡散と接着挙動に基づいて、細胞をさまざまなレジームに分類できることがわかりました。定量分析は、高密度のナノピラーアレイに播種された細胞がピラーの上に浮遊し、接着斑が平らな表面またはまばらなピラーアレイと比較して細胞周辺に近い形で形成されることを示唆しています。この変化は、柔らかい基板に播種された細胞の同様の反応に類似しています。
結果
この作業では、U2OSセルが平らなガラス表面に製造されたポリマーナノピラーのアレイによってどのように影響を受けるかを探ります。アクチン細胞骨格の組織と接着斑の位置、数、形状の変化を説明することに焦点を当てています。私たちの調査結果から、ナノピラー上での拡散と接着挙動に基づいて、細胞をさまざまなレジームに分類できることがわかりました。定量分析は、高密度のナノピラーアレイに播種された細胞がピラーの上に浮遊し、接着斑が平らな表面またはまばらなピラーアレイと比較して細胞周辺に近い形で形成されることを示唆しています。この変化は、柔らかい基板に播種された細胞の同様の反応に類似しています。
結論
全体として、ハイスループットナノファブリケーション、高度な光学顕微鏡、細胞プロセスを視覚化する分子生物学ツール、およびデータ分析の組み合わせを使用して、細胞がナノ構造表面とどのように相互作用するかを調査でき、将来、特定の誘導を引き起こす培養基質の作成に役立つことを示します細胞機能。
グラフィックの要約
背景
インビボでは、細胞は通常、細胞外マトリックス(ECM)と呼ばれる複雑な3D環境に存在します。 ECMは、細胞の構造的足場として機能するだけでなく、生体力学的および生化学的信号の伝達手段としても機能するため、組織の形態形成、恒常性、分化などのさまざまなプロセスを制御します。水、多糖類、タンパク質で構成されており[1,2,3,4]、組織の種類によって組成が異なります。
生体内の状態をよりよく表す細胞培養モデルを作成する必要性に動機付けられて、研究者はますます3Dマトリックスおよび「半3D」システムでも細胞の挙動を研究し始めています。平らな基板とより高い次元のシステムとの間の細胞表現型の多くの違いが確認されています[5、6]。たとえば、生存率、増殖、分化、形態などの特性は、平坦な表面上の細胞と3Dマトリックスに埋め込まれた細胞の間で異なることが知られています[3、7]。
インビボのような基板は、さまざまなナノ構造で装飾された平坦な表面などの「半3D」/2.5D基板から、コラーゲンゲルやマトリゲルマトリックスなどの「真の3D」システムにまで及びます[8、9、10、11]。さらに、表面上のリガンドの制御された配置は、細胞がさまざまな化学パターンとどのように相互作用するかについての新しい洞察を与える可能性があります[12、13、14]。また、構造の剛性や表面の化学的性質などの機械的要因が細胞機能に影響を与えることが示されています[15、16、17]。この目的のために、細胞研究のための多数の異なる基質が開発されてきました[3、18、19、20、21、22]。
また、3D培養システムは、薬物のin vivo効果をより正確に予測できるため、これらのシステムは創薬に応用できる可能性があることも示唆されています[16、23、24]。ナノスケールの地形パターンを正確に制御することも、細胞の形態を調節するために使用できます。たとえば、しわや溝を使用して、心筋細胞の横紋筋の整列を再現し、さまざまな疾患をモデル化するための生理学的に関連する状態をより適切に表すことができます[25、26]。
細胞の細胞骨格はECMに接続されており、接着斑(FA)、細胞表面インテグリンや足場タンパク質を含む多タンパク質複合体によって促進されます。複雑な一連の規制メカニズムに応じて、FAは、細胞移動などの前進運動に必要な回転率で形成および分解されます。 FAはECMに機械的な力を及ぼすことが知られており、逆に細胞に力を加えるECMは、膜のインテグリン親和性と結合力に影響を与えることが知られています[27]。
FAの不可欠な部分であることが知られているタンパク質の1つはビンキュリンです。これは、F-アクチンをインテグリン複合体に固定することに関与するリンカータンパク質の1つです。ビンキュリンの欠如は、細胞の形態、接着、運動性を変化させ[28]、細胞が基質に力を伝達する能力を損ないます[29,30,31]。ビンキュリンは、アクチン細胞骨格のインテグリン複合体への機械的接続に関与するだけでなく、アクチンフィラメントを架橋および束ねる[32、33、34]、既存のアクチン束を修飾する[35]、アクチンフィラメントをキャップする、新しいアクチン重合部位を核形成し[36]、アクチン修飾因子を動員します[37]。
細胞は、細胞と基質の接着の数と種類を変えることで3Dマトリックスに応答し、細胞骨格の空間構成に変化を引き起こします。これらの変化は、形成された癒着の分布、サイズ、およびダイナミクスに影響を及ぼします[4、38、39、40、41]。この再配列は、細胞増殖、形態および運動性の変化につながる可能性があります[42]。
複雑な3D環境が細胞に与える影響を理解するには、細胞プロセスを研究してフラットコントロールと比較できる新しいモデルシステムを開発する必要があります。細胞応答は培養基質の物理的、機械的および化学的特性に依存することが知られているため、正確に制御された特性を備えた細胞基質を製造することが望ましい[43、44、45]。さらに、光学顕微鏡などのすでに確立された分析技術を使用して、細胞と基質を簡単に研究できる場合は非常に有利です。
最近注目を集めている基板の1つは、ナノピラーまたはナノワイヤーで装飾された平坦な表面です[18、21、46、47、48、49、50、51、52、53]。たとえばヒドロゲルと比較すると、これらの構造化された表面は真の3D環境を模倣していませんが、明確に定義された表面トポグラフィーを持っています。これらの基板は通常、2.5Dと呼ばれます。このようなシステムは、生物学的に関連する分子の細胞への送達を促進し[54、55]、酵素活性を監視し[56]、核力学をテストし[57]、膜の曲率の調整がさまざまな細胞膜関連にどのように影響するかを研究するためにすでに適用されています。プロセス[58,59,60]。透明な基板上にナノ構造を作製することにより、このアプローチを光学顕微鏡と統合することが可能です。
さまざまな細胞株、ナノ構造のタイプ、および形状の可能な組み合わせの数は多く、文献からの例は豊富です。 Li etal。ランダムに配置されたリン化ガリウムナノ構造で装飾された表面での細胞の挙動を説明し、大きなFAを持つ細胞の割合を定量化しました[61]。細胞とFAの形態は、ナノワイヤのさまざまな面積密度の表面で調査されました。結果は、低密度の表面に播種された細胞が基板と接触しており、細胞の端の周りに大きなFAを形成したことを示した。これらのアレイのセルの大部分で大きなFAが検出されました。ナノワイヤの面密度が高い場合、細胞はナノワイヤアレイの上部に浮遊し、細胞の下に点状のFAが観察されました。これらのアレイ上の細胞の割合が低いと、ナノワイヤの面密度が低い表面上の細胞と比較して、大きなFAが示されました。
Buch-Månsonetal。 Si基板上にランダムに配置されたシリコンナノカラムアレイのセルナノ構造表面相互作用を研究しました[62]。使用される製造プロセスでは、面密度は制御されましたが、ピラー間の距離は制御されませんでした。 FAの調査は、中間の面密度を有するアレイ上のセルが、最も非対称な形状を有するFAの数が最も多いことを示した。これらのFAのいくつかがナノカラムの側壁に形成されたことが示唆された。これは、面積ナノカラム密度が低い表面と高い表面では観察されませんでした。
以前の研究では、平らなガラス表面上にSU-8ポリマーナノ構造を製造するための詳細なプロトコルを説明し[63]、これらの表面上の2つの異なる細胞株の細胞挙動を調査しました[45、48]。この作業では、電子ビームリソグラフィー(EBL)を使用して、垂直に整列したSU-8ポリマー構造で装飾された表面を作製し、骨肉腫上皮細胞株U2OSのアクチン細胞骨格およびFA組織の変化を研究します。さまざまなトポロジー的手がかりを使用して、表面によって引き起こされる変化の定性的分析と定量的分析の両方を実行します。
結果
以前に確立されたプロトコルを使用して、可変分離と定義された形状を持つ垂直配向SU-8ナノピラー(NP)の正確に定義されたアレイで装飾されたガラスカバースリップを製造しました[63]。 NP面密度が456、205、115、および29 NP /100μm 2 の表面 (500 nm、750 nm、1000 nm、2000 nmのピッチに対応)が使用されました。まず、細胞の形態、アクチン細胞骨格の構造、細胞と基質の相互作用の一般的な傾向を調べます。これに続いて、さまざまなナノ構造基板と板ガラスコントロール上の細胞とFAの形態を定量的に比較します。高解像度顕微鏡とハイスループット製造を組み合わせて、各表面タイプについて少なくとも\({\ approx 100} \)細胞の定性分析を行い、24時間後と48時間後にイメージングを行います。合計で、400を超える高解像度画像と20の3Dデータセットを分析します。
図7は、NPアレイの概略図(A、B)と、作製した基板の電子顕微鏡画像(C、D)を示しています。ナノ加工された構造を含むスライドガラスは、構造が上を向くように、35mm培養の中空底の下にパラフィンを使用して取り付けられました。図1C、Dに示すトップダウンのタイトル付きサイドビュー電子顕微鏡画像は、ピッチと高さが1000nmのナノピラーアレイを示しています。表1は、この作業で使用されるアレイの幾何学的パラメーター、それらの分類、および対応するNP面積数密度を示しています。観察された細胞接着挙動に応じて、NPアレイを高密度と疎に分類します(以下を参照)。
<図>図1は、ガラス(A)とナノ構造表面(B–F)で24時間培養された代表的なU2OS細胞を示しています。細胞はpCMV-LifeAct-GFPとpTAG-RFP-Vinculinでコトランスフェクトされており、蛍光性のLifeAct-TagGFP2(以下:LifeActGFP)とTagRFP-ビンキュリン融合タンパク質の生成を通じてF-アクチンとビンキュリンをそれぞれ可視化できます。 FAのF-アクチンネットワークとビンキュリンが豊富な領域が明確に検出されます。細胞周辺では、LifeActGFPシグナルが膜に近接しているため、このシグナルを使用して細胞の形態を視覚化します。 SU-8 NPからの信号は青色で示されています(「実験」セクションを参照)。
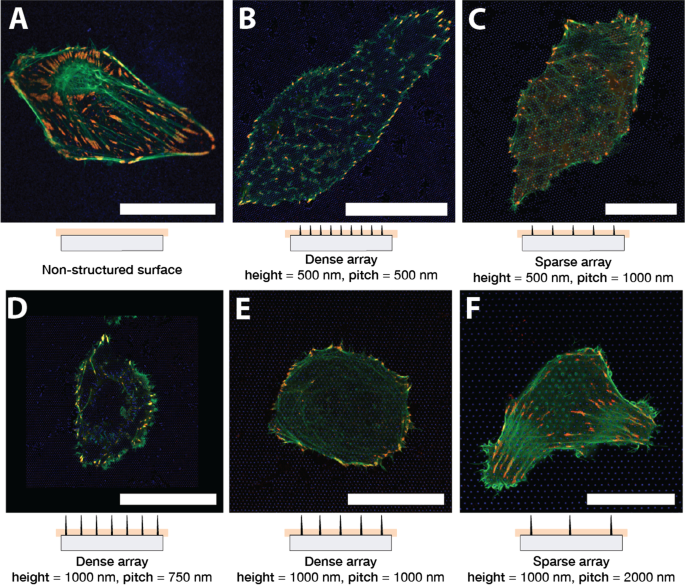
異なる表面タイプで蛍光LifeActGFP(緑)とTagRFP-ビンキュリン(赤)を発現するU2OS。黄色は、LifeActGFPおよびTagRFP-ビンキュリンチャネルからの信号が重複していることを示します。各顕微鏡写真の下に、対応するNPアレイの概略側面図が、取得平面のおおよその位置とともに示されています。 A で画像化された細胞 非構造化された板ガラス表面と、アレイのピッチと構造の高さが異なるピラーアレイ: B ピッチ500nm、高さ500 nm、 C ピッチ1000nm、高さ500 nm、 D ピッチ750nm、高さ1000 nm、 E ピッチ1000nm、高さ1000 nm、 F 2000nmのピッチと1000nmの高さ。提示されたすべての画像は代表的な細胞のものです。スケールバー25μm。 C に注意してください 他の画像とはスケールが異なります
トランスフェクトされた細胞を使用した予備テストでは、ガラスと構造化された表面の両方に播種された細胞が約6時間後に完全に広がっているように見えました。異なる表面間での細胞の広がりに明らかな違いは観察されず、目視検査では、この時点または後の実験でピラーアレイによる細胞生存率の低下の兆候は見られませんでした。次の実験では、細胞を播種の6時間後にトランスフェクトし、トランスフェクションの24時間後、48時間後に画像化しました。これは、播種後30時間、54時間のトランスフェクションに対応します。以下では、これら2つの時点を、播種後の観察時間、つまり24時間、48時間で参照します。
高さ500nm、1000 nm、ピッチ750 nm、1000 nm、2000nmのNPアレイに細胞を播種しました。最初の拡散後、平らな表面の状況と同様に、細胞は円形または細長いかのいずれかであることが観察されました。この一般的な形態は、複数の実験にわたって一貫していることがわかりました。スパースNPアレイに播種された細胞は、通常、ガラス表面の細胞に似た形状でした。2000nmピッチのアレイ上の代表的な細胞を示す図1Fを参照してください。 F-アクチン繊維は、NPのベースとガラス表面の近くにも存在し、細胞が基板に近い領域にアクセスできたことを示しています。以前に観察されたように[45、62、64]、高密度アレイ上の細胞は通常、NPの上に浮遊しているように見えました(図1B、D、E)。高密度アレイ上の細胞は、ガラス表面の近くにあまり目立たないF-アクチンを持っているように見えます。これは、アクチン繊維が基板に近い柱の間に形成されなかったことを示しています。 NPの高さと分離の関係は、細胞が基板に付着したのか、NPアレイの上部に浮遊したのかを決定しました。これは、例えば、図1Cに示され、短いNPは、細胞が基質に接触することにつながるが、長いNPは、接触を妨げた、図1E。アクチン繊維のこれらの観察は、図2に示すように、いくつかの表面に対して実行されたzスタックによってさらに裏付けられました。
細胞が構造化および非構造化表面にどのように付着するかについてより詳細な理解を得るために、TagRFP-ビンキュリン融合タンパク質の存在によって視覚化されたFAの分布を評価しました。図1Aに示すように、平らな表面の細胞は通常、細胞体全体の下に分布する細長いFAを形成しました。
スパースアレイでは、U2OSはNP間のガラス表面に接触し、図1C、Fに示すように、ガラス上の細胞と同様に接着することができました。これらのNPアレイでは、NP間のガラス上に形成されたFAとF-アクチンシグナルNPのベース近くで取得された画像でも検出されました。これは、細胞がナノ構造の周りで膜を曲げることができたことを示しています。ただし、750 nm、1000 nm nmの間隔、1000 nmの高さなど、より高密度のアレイ上の細胞は、図1D、Eの基部近くで取得された画像に示すように、ナノ構造間の基板への付着が明らかに妨げられていました。 NP。ただし、周辺では、細胞は通常、FAを形成するナノ構造間の基板に付着することができ、多くの場合、下にあるピラーアレイの対称性によって誘導されます。
長さが短く、ピラー間の間隔が1000 nmのNPアレイ上に広がる細胞は、細胞体の周囲と下の両方に癒着を形成しました。 F-アクチン繊維の配向は、図1Cに示すように、下にあるアレイの対称性によって方向付けられました。ただし、FAを含むビンキュリンの位置と方向は明確なパターンを示さず、FAはNPの間に形成されました。
図1Bに示すように、ピラー間距離が500nmの500nmピラー上のU2OSセルは、一般に、平面に比べて癒着が少なくなり、小さくなります。このアレイに播種された細胞の場合、アクチン繊維は主に、FAで終端する位置でガラス表面の近くで観察されました。繰り返しますが、これはアクチンネットワークが表面に接触するのを妨げられたという兆候であり、したがって細胞はアレイの上に浮遊していると想定されました。しかし、細胞は柱の上に形成されたより無傷のF-アクチンネットワークを持っているように見えました。柱の間で観察されたアクチンネットワークの一部は、下にあるNPアレイの構造と整列しているように見えました。これは図1Bで見ることができます。これは、F-アクチン繊維とFAが主に、ピラーアレイの格子方向の1つに沿って、つまり開いた「線」に平行に形成されるためです。
密な配列と疎な配列の両方で、細胞体に上向きに突き出ているNPの周りに形成された「リング状」のF-アクチン構造を観察しました。図1E、Fに示すように、F-アクチンリング構造はスパースアレイでより顕著であるように見えました。
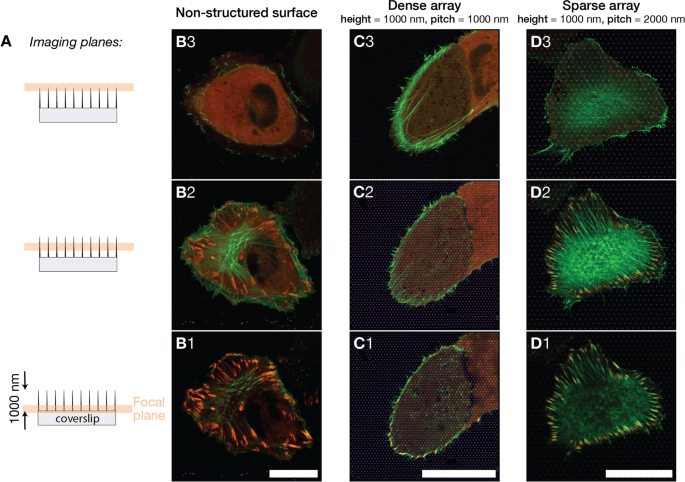
A 異なる表面上のU2OS細胞の画像は、3つの示された焦点面で取得されました。画像は、柱の基部の近く、ほぼ半分の柱の高さ、および柱の頂点の近くで取得されました。イメージング平面は、平らなガラス表面から0.0 µm、0.4 µm、0.8 µmの位置にありました。 B – D )異なる表面タイプで蛍光LifeActGFP(緑)とTagRFP-ビンキュリン(赤)を発現するU2OSを示すマージされた蛍光画像。黄色はLifeActGFPとTagRFP-ビンキュリンの重複を示します。画像化された表面は B 非構造化ガラス表面、 C 1000 nmピッチのピラーアレイ、 D 2000nmピッチのピラーアレイ。提示された画像は、各表面タイプの細胞を代表するものです。各表面タイプの焦点面間の垂直距離は約400nmです。スケールバー25μm
上記の結果に基づいて、細胞の形態とFAをより詳細かつ定量的に説明するために3つの表面を選択しました。密なアレイ(ピッチ1000 nm、長さ1000 nm)、疎なアレイ(ピッチ2000 nm、長さ1000 nm)のセルを調査し、その結果をコントロールとして使用される平らなガラス表面のセルと比較します。
Airyscan検出器を専用の画像後処理と併用することにより、約140nmのxy解像度と約400nmのz解像度でイメージングを実行することができました[65]。図2は、3つの表面上の細胞の画像を示しています。画像平面は約400nm離れています。さまざまな z でのF-アクチンバンドルの調査 。平らな表面の細胞は、同じ焦点面またはFAの真上にはっきりと見えるF-アクチンネットワークを持っていました(図2-B2を参照)。高密度アレイ上のセルの場合、F-アクチンネットワークは、ガラス支持体と接触していたFA平面と比較して、セル内のより高い焦点面で見つかりました(図2-C1およびC2 / C3)。スパースアレイ上のセルの場合、状況はガラスコントロールとアクチンネットワーク上のセルと同様であり、FAは同じ高さで検出されました(図2-D2)。このデータは、疎なアレイ上の細胞が構造間の表面に付着したのに対し、密なアレイ上の細胞は主に細胞周辺の表面に付着することができたという最初の観察を裏付けています。
選択した3つの表面のFAと細胞形態の違いを分析および定量化するために、Pythonベースの画像分析スクリプトを使用しました(「実験」を参照)。定量分析のために、300以上の高解像度画像が分析されました。これらの画像では、> 400個のセルと> 7700個のFAが識別されました。表2に、分析に含まれる3つの表面タイプで検出されたセルとFAの数を示します。すべての表面について、トランスフェクションの24時間後と48時間後の両方で細胞を画像化しました。次の分析では、3つの表面タイプと24時間後、48時間後のセルの表面積、真円度、アスペクト比などの幾何学的パラメータが比較されます。追加の分析は、補足情報にあります。セルの表面積、真円度、アスペクト比を図3に示し、FAの数、セルあたりのFA面積の合計、およびセルに対するFA面積の割合を図4に示します。幾何学的パラメーターは「実験」で説明したように定義しました。セクション。
<図>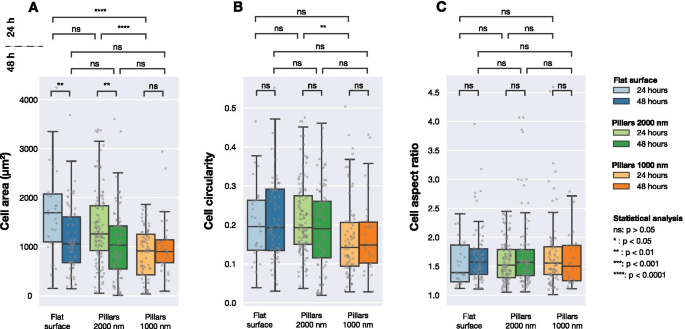
トランスフェクションの24時間後と48時間後に画像化された、さまざまな表面で蛍光LifeActGFPおよびTagRFP-ビンキュリン融合タンパク質を発現するU2OS細胞の計算された細胞面積、細胞円形度、および細胞アスペクト比。画像検出は、LifeActGFP信号の強度に基づいて実行されました。各灰色の点は1つのセルに対応し、箱ひげ図には中央値( Q )が表示されます。 2)および最初の( Q 1)および第3四分位数( Q 3)。分布間の統計的差異は、マン・ホイットニーのノンパラメトリック検定を使用して評価されました
図3は、平面と構造化表面の両方について収集されたデータをまとめたものです。図3Aに示すように、24時間の細胞培養後、細胞面積に有意差が観察されました。ただし、48時間後、調査した表面の平均セル面積に有意差はありませんでした。細胞の真円度を考慮すると(図3B)、24時間後に画像化された密な柱と疎な柱に播種された細胞を除いて、異なる表面間で有意差は検出されませんでした。図3Cに示すように、3つの表面すべてのセルの平均アスペクト比は同じでした。
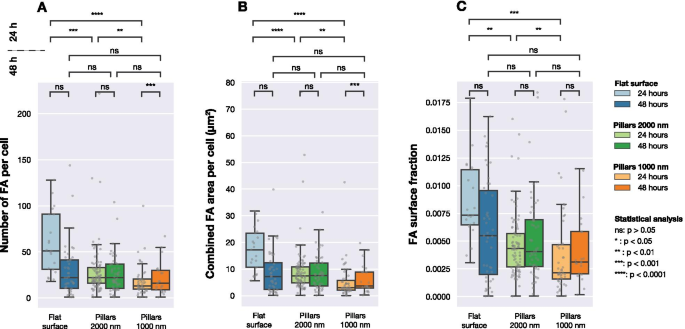
FAの数、細胞あたりのFA面積の合計、および3つの異なる表面タイプ(1000nmおよび2000nmのピッチのフラットおよびピラーアレイ)で画像化された細胞の細胞面積に対するFA面積の割合。各灰色の点は、1つのセルからの観測に対応します。分布間の統計的有意性は、Mann-Whitneysノンパラメトリック検定を使用して決定されました
図4は、セルごとに検出されたFAの数の分布、各セルのFAの総表面積、およびセル面積に対するFA面積の比率を示しています。 24時間後、3つの異なる表面の細胞によって形成されたFAの数は大幅に異なりました。図4Bに示すように、細胞あたりの総FA表面積は、平らな表面と構造化された表面に播種された細胞で異なっていました。図4Cに示すように、さまざまな表面のFAの相対量(検出されたFAの総面積を総セル面積で割ったもの)を比較した場合にも同じことがわかります。
ただし、48時間の培養後、細胞集団間の有意差は観察されなくなりました。セルあたりのFAの数、セルあたりのFA面積の合計、またはFA表面の割合を考慮すると、3つの表面の間に違いは見られませんでした。
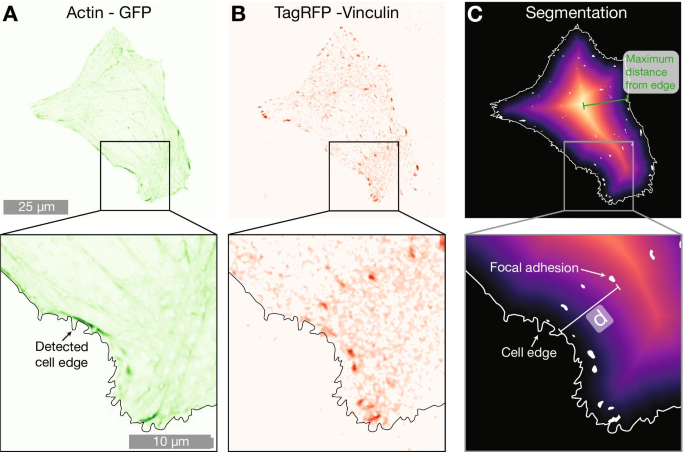
A を表現する2000nmピラーアレイ上のセルの例 LifeActGFP(緑)と B TagRFP-ビンキュリン(赤)。両方の図に重ねて表示されているのは、LifeActGFPの発現からのシグナルを使用して決定された検出された細胞エッジです( A に示されています)。 )。 C 検出されたセルエッジと検出されたFAからの距離マップを表示します。最短距離( d で示される) 図では、セグメント化された各ビンキュリンスポット(白い領域)から細胞周辺(白い実線で示されている)まで、画像内のすべてのFAについて計算されました
NPの存在が細胞内のFAの局在に影響を与えるかどうかを理解するために、FAの位置を使用してさらに分析を行いました。顕微鏡データは、高密度NPアレイ上の細胞内のFAが、図1および2に示すように、細胞周辺に近い位置にあることを示しました。この傾向を定量化するために、各FAからセルエッジまでの最短距離を計算しました。これは、図5に示すように実行されました。F-アクチンを使用して周辺の位置を決定し、距離マップを作成することにより、検出されたFAの各中心からセル周辺までの距離を計算しました。セルサイズの違いを説明するために、検出されたセルエッジとFAの間の距離を、各セルのエッジから幾何学的中心までの最大距離(円形のセルの半径に相当する距離)で正規化しました。最大距離で正規化されたデータを図6と表3に示します。平面上のセルの場合、FAはセルの中心に向かってより多く分布しますが、スパース2000nmアレイとデンス1000nmアレイの両方でFAはセル周辺に近い位置にあります。この効果は、1000nmピッチの高密度アレイで最も顕著です。 FAの位置が細胞の表面積によって正規化された代替の正規化アプローチの結果は、補足情報に含まれています。このデータは、図6に示されている各セルのエッジまでの最大距離で正規化されたデータと同じ定性的な傾向を示しています。
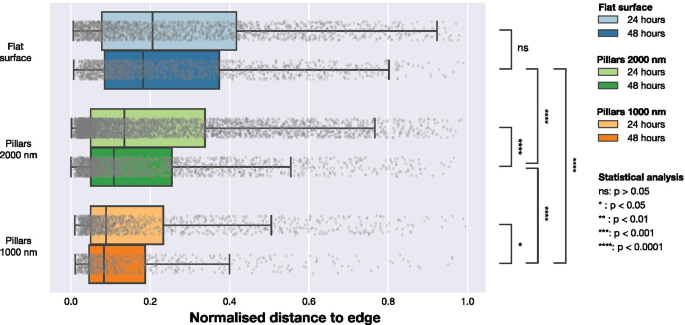
セルの幾何学的中心からエッジまでの最大距離で正規化された、最も近いセルエッジに対するFA位置の分布。距離は、観測された各FAからLifeActGFP信号によって定義されたセルエッジまでの距離を計算することによって取得され、24時間と48時間で3つの表面タイプについてプロットされました。灰色の点はFAの個々の観測値を表し、それらの分布は箱ひげ図にまとめられています。異なる表面と時点からのFAが同じ分布からのものである可能性をテストするために、図に示されている有意水準ですべての分布に対してマンホイットニー検定が実行されました
ディスカッション
アクチン細胞骨格の組織化と癒着の形成は、平らな表面で広く研究されているプロセスです。本研究は、アクチン細胞骨格の組織化とナノピラーアレイ上の接着斑の変化を調査および定量化するために設計されました。この研究では、ナノピラーアレイが細胞移動にどのように影響するかについては調査していません。ただし、胚性マウス線維芽細胞の同じピラーアレイで以前に観察されたように、高密度アレイ上の細胞はより高い運動性を示す可能性があると予想されます[45]。
細胞が拡散して表面に長時間付着することを可能にすることで、アクチン細胞骨格の組織化と完全に成熟したFAの存在を観察することが可能になります。播種から24時間後、表面に播種された細胞の細胞面積、真円度、アスペクト比に大きな違いが見られます。ただし、48時間後には有意差は検出されませんでした。これは、24時間後の細胞がまだ表面に完全に付着しておらず、ナノ構造が完全に成熟する前に主にFA組織に影響を与えることを示しています。 24時間後も48時間後も、NPの上部または側面にFAが形成されることはありません。
アクチン細胞骨格組織の変化は、細胞が周囲とどのように相互作用するかも関係しています。たとえば、ストレスファイバーとFAの両方は、ストレッチを受けると成長し、機能的に相互依存しているように見えます[66]。他の報告によると、柔らかい表面または順応性のある表面に播種された細胞では、より丸い細胞とエッジ周辺のFAの局在がしばしば観察されます[67]。私たちの結果では、同様の傾向が見られます。密集したアレイ上のセルは、図1および2に示すように、セルの端の周りに繊維を示す傾向があります。 1Eまたは2B。 FAは、これらのセルのセルエッジの近くに形成されているように見えます。細胞が柱の上に吊るされているときのように、細胞が平らな表面を欠いているとき、FAの分布はPrager-Khoutorskyや同僚によって使用されているような柔らかい基板上のFAに似ていると推測します[67]。
>私たちの結果から、柱の上に浮遊している細胞は、柱の上に発達したアクチンネットワークを持っているように見えますが、柱自体にFAが形成されていないことがわかります。ただし、スパースアレイ上のセルの場合、セルはNPの影響を受けにくく、アクチンネットワークとFAの両方がより「平坦な表面のよう」に見えます。
FAとアクチン細胞骨格の間の相互作用は複雑であり、まだ完全には特徴付けられていません。アクチン細胞骨格をECMにリンクするFAは、牽引点として機能し、細胞内のストレスファイバー形成を促進することが知られています。逆に、アクチン繊維は再びFAの組織化と成熟に影響を与えています。多くの研究は、細胞が高密度のNPアレイの上にどのように浮遊する傾向があるか[61、62、68]、および細胞膜が単一のNPとどのように相互作用するか[69、70、71]を説明しています。これらの観察結果は理論的研究[64]によって裏付けられており、柱の細胞の挙動はかなりよく理解されています。
FAの形成と高密度アレイ上のセルエッジ周辺の基板への付着の背後にあるメカニズムは不明なままです。この点で、柔らかい基板上のセルとの比較は特に興味深いものです。 For soft substrates, actin fibres are organised in a ring like fashion close to the cell edge and FAs form around the cell periphery [67]. On the nanopillar arrays similar type of architecture is observed, but the actin fibres are typically shorter. Similar qualitative trends in terms of actin organisation and FA formation were observed by Li et al. for cells seeded on random nanowire arrays made from gallium phosphide [61].
In our studies we also observed formation of F-actin rings around NPs. The formation of F-actin rings around NP has previously been described for fibroblasts on similar surfaces [45] and for U2OS cells on nanostructures with a range of structure sizes [58].
Contrasting our results to other studies highlight an important aspect of studies on cellular response to NP arrays:cellular response may vary considerably depending on cell type, NP material, NP geometry and as well as other parameters. For example, Buch-Månson et al. studied fibroblasts and investigations of FAs showed that cells suspended on arrays with intermediate NP density had the highest number of FAs. In our results we do not see a similar trend. However, these studies cannot be directly compared as Buch-Månson et al. studied another cell line using a system with different array geometry, surface porosity and NPs length [62].
There are also studies describing the effect FAs placement has on cells [41]. By modelling cells on planar substrates Stolarska et al. suggest that the cells can control intra-cellular stresses by three mechanisms:FA position, FA size and attachment strength. FAs around the periphery allows the cells to be more sensitive to changes in the micro-environment. This could also be an underlying mechanisms for cells on NPs. Yet, it is not obvious that the results for the planar substrate are directly transferable to NP decorated surfaces.
Cell-interactions with the surrounding environment, for flat substrate, NPs arrays or in vivo ECM, are regulated by a complex set of relations between actin organisation, membrane mechanics, cell dynamics and contact with FAs. To further explore these relations, applying flat surfaces structured with NPs could be one promising approach. Such surfaces may also aid in exploring discrepancies in the cellular response to environmental cues between different cell lines.
結論
In order to create more physiologically relevant systems for cellular studies, a plethora of 3D and 2.5D approaches have been proposed. One approach is to use flat-surfaces decorated with vertically aligned nanostructures as a simple model system. High resolution live cell imaging of co-transfected U2OS cells expressing pCMV-LifeAct-GFP and pTAGRFP-Vinculin have been used to study the influence of nanopillar arrays on actin cytoskeleton focal adhesion organisation. Our present results indicate that the U2OS cells spreading on surfaces decorated with nanopillars can be categorised into three different regimes by how they respond to the nano-structures. These observed changes are quantified by analysing more than 400 high-resolution images, and indicate that tuning geometrical properties of the nanostructured surface can be used to direct cell behaviour.
More specifically, the U2OS cells were found to either contact the substrate, attach preferably around the cell edge, or be fully suspended on top of the vertical NP arrays. In the latter case, we hypothesise that the resulting reorganisation of FA and cytoskeleton is an effect analogous to what is seen for softer substrates.
Increased understanding of how cells behave on nano-structured surfaces, such as pillar arrays, could help us discover more details about complex cellular processes. For example, it is still poorly understood how changes in the actin cytoskeleton and its architecture influence cell signalling. By studying the cell response on nanostructured surfaces in a systematic way, the potential connection between actin cytoskeleton, cell adhesions and a plethora of biochemical signalling pathways could be further explored. We therefore envision that further development of the presented platform and analysis could have implications for advanced in vitro applications or for development of smarter in vivo biointerfaces.
メソッド
Fabrication of Nanostructures and Sample Mounting
SU-8 nanostructures were fabricated as previously explained [63]. Briefly, 24 mm by 24 mm glass cover slips (#1.5, Menzel-Gläser, thickness 170 μm) were cleaned by immersion in acetone, isopropyl alcohol, rinsed in de-ionised water and dried. The cover slips were then oxygen plasma treated for 2 min (Diener Femto plasma cleaner, power 100 W, base pressure 0.3 torr), followed by dehydration for 10 min on a 150 °C hot plate. Samples were then placed in a desiccator containing an open vial of Hexamethyldisilazane (HMDS, Sigma Aldrich product no:440191). HMDS was applied by vapour deposition, the desiccator was pumped to low vacuum using a diaphragm pump for 5 min and the samples were kept in HMDS atmosphere for 60 min.
Substrates for EBL were prepared directly after HMDS treatment by spin coating SU-8 2001 (Microchem Corp.) to a desired thickness of 500 nm and 1000 nm. SU-8 was made fluorescent by adding either Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102 (all Sigma Aldrich) to a final concentration of 100 μg mL −1 resist. After spin coating samples were dehydrated on a hot plate at 95 °C. To mitigate charging during EBL exposure samples were then covered by a layer of conductive polymer AR-PC 5091 Electra 92 (AllResist GmbH) by spin coating at 2000 rpm for 60 s to thickness of 50 nm.
An Elionix ELS-G100 100 kV EBL-system was used to fabricate SU-8 nanopillars (NPs) with processing parameters as described in our previous work [63]. Table 1 summarise the arrays fabricated for this work. Pillar arrays were exposed using the Elionix dot-pattern generator where each pillar is exposed in a single exposure. Arrays were exposed over an area of 2000 μm by 4000 μm, with a current of 500 pA in write fields of 500 μm by 500 μm. NPs had a tip diameter of about 100 nm as a base diameter of 150 nm and 200 nm for structures of length 500 nm and 1000 nm respectively.
After EBL exposure, the samples were rinsed in DI-water to remove the conductive polymer, then post exposure baked for 2.3 min at 95 °C and developed twice in mr-Dev 600 (Micro Resist Technology GmbH) developer for 20 s, rinsed in isopropyl alcohol and dried. Samples were then treated with oxygen plasma (Diener Femto plasma cleaner, power 50 W, base pressure 0.3 torr) for 30 s to render SU-8 hydrophilic and to give it similar surface chemistry as glass by oxidising surface epoxy-groups to hydroxyl.
Fabricated structures were imaged using Scanning electron microscopy (SEM) and samples sputter coated with 5 nm Platinum/Palladium alloy deposited with a 208 HR B sputter coater (Cressington Scientific Instruments UK). SEM was performed with a FEI Apreo SEM, at 5 kV and 0.2 nA with sample 45° pre-titled stage and with additional tilting of 30°.

A Side view schematic representation of nano-structured surface mounted in petri dish. Glass slides are mounted using paraffin such that structures are pointing upwards. B Tilted schematic representation of nano-pillar array on flat surface, and two important parameters for the nano-pillar arrays (height and pitch). These figures are not drawn to scale. C , D Overview of the nanopillar arrays employed in this work. Top-down and tilted side-view scanning electron micrographs of fabricated nano-pillar array with pillars of height 1000 nm and pitch 1000 nm. Scalebars 2000 nm
When exposing the pillars, an indexing system was also exposed to make navigation during live-cell imaging more reliable. Arrays were optically inspected after fabrication to ensure free and standing pillars. The short Oxygen plasma treatment to render the SU-8 structures did not lead to any optically visible change to the structures. Lastly, the samples were mounted underneath 35 mm diameter dishes (Cellvis, Mountain View, CA, USA) with 14 mm holes and nano-structures pointing upwards, as indicated schematically in Fig. 7. As flat surfaces, areas outside the structured part of the same samples were used. Before usage, all dishes were disinfected with 70% ethanol twice and dried.
Cell Culture and Transfection
U2OS-cells (ATCC) were cultivated in Dulbecco’s modified Eagle’s Medium (DMEM Prod. 41965039, Fischer Scientific) with 10% fetal bovine serum (FBS) and kept at 5% CO2 and 37 °C. Before detachment, cells were washed with PBS and detached with Trypsin-ethylenediaminetetraacetic acid (trypsin-EDTA) and seeded on nanostrucutred or flat surfaces. For the diameter 14 mm glass wells 15,000 cells were seeded.
For the standard transfection experiments, cells were allowed 6 h for adhering to surfaces before transfection. U2OS cells were transiently transfected using Lipofectamine 2000 (Invitrogen, Fischer Scientific) by adapting the manufacturer protocol to our system. Briefly, 2 μL Lipofectamine 2000 was added to 50 μL Opti-MEM I Reduced Serum Media (Prod. 11058021, Gibco , Fischer Scientific) and incubated for 5 min at room temperature. Plasmid DNA coding for fluorescent LifeAct-TagGFP2 and TagRFP-vinculin fusion proteins were co-transfected by using 0.5 μg plasmid DNA (vinculin-pTagRFP and pCMVLifeAct plasmids) was diluted in 50 μL Opti-MEM I and incubated at room temperature for 5 min. For co-transfection of TagRFP-vinculin and pCMVLifeAct 0.5 μg of each plasmid was used.
The diluted DNA was added to the diluted Lipofectamine 2000 in a 1:1 ratio, and left to incubate for 20 min at room temperature. 40 μL of the combined transfection complex was then added to each well. After 18 h, 1.5 mL DMEM (Prod. 41965039) supplemented with 10% FBS and 1% 10000U/mL Penicillin-Streptomycin was added to each dish.
For reverse transfection experiments, the same amounts of reactants were used, but the transfection complex was added to a suspension of U2OS cells, and the suspension was then added to the wells.
Microscopy
Live cell imaging was performed usin g a Zeiss LSM 800 Airyscan with an inverted Axio Observer Z1 stand connect to a PeCon compact incubator. Imaging was performed in an humidified environment at 37 °C, with 5% CO2 flow. High resolution imaging was performed using a Zeiss Plan-Apochromat 63x/1.4NA DIC M27 oil objective with Cargille Immersion Oil Type 37 (n =1.51) suited for use at 37 °C. All images were taken using the system optimised pixel size both in-plane (typically 34 nm) and for stacks in the vertical axis (typically 180 nm).
To minimise imaging bias, imaging was performed in a standardised manner where each pillar array was raster scanned and cells expressing both LifeActGFP and Vinculin RFP were imaged. The high resolution images were then processed using a Zeiss algorithm for reconstruction of AiryScan images and exported as CZI-files for further manual and automatised image processing.
Image Analysis
For all cells, cell shape was based on the expression LifeActGFP fusion protein and expression of TagRFP-vinculin was used to identify FAs. Segmentation of images was performed using a script written in Python 3 [72] using CZIfile [73] (version 2017.09.12) for reading the microscopy images in Zeiss-format. The python packages Scipy [74] and Scikit-image [75] were used for multi-dimensional image processing and image segmentation respectively.
To reduce the influence from fluorescence cross-talk from pillars (due to Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102), the pillar/surface channel was used as a background and subtracted from the TagRFP-vinculin imaging channel. A median filter (size:10 pixels) was applied to remove noise from the TagRFP-vinculin channel, followed by classification of the image into regions based on their intensity value using a Multi-Otsu approach. Multi-Otsu thresholding with three classes was applied. The first class was typically the background, the second class constituted the cytosolic vinculin, whereas vinculin rich areas in FAs appeared brighter and could be classified into a third class. The quality of the image segmentation was briefly assessed by comparison to manual segmentation.
Area of cells and vinculin rich regions were described by counting pixel numbers and from this the actual area was found by correcting for the pixel size. Shape geometries were described by fitting each region with an ellipse with the same second-moment as the segmented region. In order to describe the cell area geometry, three measures were used:(1) Aspect ratio defined as the ratio of the ellipse major axis to the minor axis. (2) Circularity given as,
$$\begin{aligned} C =\frac{4\pi *\text{Area}}{\text{Perimeter}^2}, \end{aligned}$$ (1)and roundness given as,
$$\begin{aligned} R =\frac{4*\text{Area}}{\pi *\text{MajorAxis}^2}. \end{aligned}$$ (2)Segmented vinculin areas with a fitted ellipse that were too round (aspect ratio \(\le 1.5\)) or too elongated (aspect ratio \(\ge 8.5\)) were rejected. In addition, vinculin areas smaller than 0.05 μm 2 were filtered out. In order to find the distance between each vinculin area and the cell edge, the shortest euclidean distance between each centroid (the centre of the fitted ellipse for each vinculin area) and the cell edge was calculated.
Statistical Analysis
Statistical comparisons of distributions were performed by using the non-parametric two-tailed Mann-Whitney test neither assuming normal distribution nor equal standard deviation. P -values \(\ge {0.05}\) were considered to represent a non-significant (ns) difference between the two populations. Significant values were denoted with * for p in 0.01 to 0.05, ** for p in 0.001 to 0.01, *** for p in 0.0001 to 0.001 and lastly **** for p \(\le {0.0001}\).
略語
- DMEM:
-
Dulbecco’s Modified Eagle’s Medium
- EBL:
-
Electron Beam Lithography
- ECM:
-
Extracellular Matrix
- EDTA:
-
Ethylenediaminetetraacetic Acid
- FA:
-
Focal Adhesion
- FBS:
-
Fetal Bovine Serum
- GFP:
-
Green Fluorescent Protein
- HMDS:
-
Hexamethyldisilazane
- NP:
-
Nanopillar
- RFP:
-
Red Fluorescent Protein
- SEM:
-
Scanning Electron Microscopy
ナノマテリアル



