ポリスチレン-co-アクリル酸複合ナノスフェアを用いたコレラ菌DNAの検出のための超高感度バイオセンサー
要約
病原性の Vibriocholerae を測定するための超高感度電気化学バイオセンサー ( V 。 コレラ )DNAは、ポリスチレン-co-アクリル酸(PSA)ラテックスナノスフェア-金ナノ粒子複合材料(PSA-AuNPs)DNAキャリアマトリックスに基づいて開発されました。電気活性アントラクニノンオリゴヌクレオチド標識を使用する示差パルスボルタンメトリー(DPV)を使用して、バイオセンサー応答を測定した。 DNA-ラテックス粒子電極への金ナノ粒子(AuNPs)のロードは、DNAハイブリダイゼーションのファラデー電流を大幅に増幅しました。報告されたプローブの使用とともに、バイオセンサーは高い感度を示しました。 DNAバイオセンサーは、1.0×10 -21 のターゲットDNAに対して再現性のある広い線形応答範囲をもたらしました。 〜1.0×10 -8 M(相対標準偏差、RSD =4.5%、 n =5)検出限界(LOD)が1.0×10 -21 M( R 2 =0.99)。バイオセンサーは、91〜109%( n )の満足のいく回復値を取得しました。 =3) V の検出用 。 コレラ スパイクされたサンプル中のDNAであり、再現性RSD値が5%( n )の6つの連続したDNAアッセイに再利用できます。 =5)。電気化学的バイオセンサーの応答は安定しており、58日間の保管期間まで元の応答の95%で維持可能でした。
背景
コレラ菌 、食品媒介性病原体は、急性水様性下痢の状態を介して、ヒトにコレラの流行を引き起こす可能性があります。コレラの発生は、世界の一部の地域では依然として深刻な問題です。アジアとアフリカ、そして低い社会経済的地位につながる[1,2,3,4]。この腸内病原体は、特に発展途上国において、罹患率と死亡率の主要な原因です[5]。さまざまな地域でのコレラの流行とパンデミックは、主に Vによって引き起こされます。コレラ 血清型O1およびO139 [1、2、6]。 V。コレラ 血清型O1には、2つの主要な血清型、すなわち稲葉と小川があり、これらはコレラの流行を交互に繰り返す可能性があります。 3番目の血清型である彦島も存在しますが、まれで不安定です。 O1抗原生合成に関与する遺伝子はrfbと呼ばれています。血清型稲葉と小川を定義する突然変異は、rfbT遺伝子の単一の欠失突然変異です[7]。しかし、重度の下痢を患うヒトにおける時折の食品媒介性の発生は、非O1 /非O139 Vによって引き起こされることも報告されています。コレラ 調理が不十分なシーフードの摂取[8]または汚染された水生環境への曝露[9]による。 Vの最初の流行。コレラ O139は1992年にバングラデシュとインドで発生し、その後東南アジアの他の国々に急速に広まりました[10]。世界中で、2005年に合計131,943件のコレラ症例と2272人の死亡が世界保健機関(WHO)に報告されました[11]。
毒素産生性 Vのモニタリングまたは診断のための効果的な方法の探求。コレラ コレラの流行を制御するには、バクテリアが不可欠です。 Vの従来の識別。コレラ 多くの場合、細菌の分離とスクリーニングによって達成されます。この場合、アルカリペプトン水(APW)での事前濃縮と、それに続く Vの分離が含まれます。コレラ チオ硫酸クエン酸胆汁塩スクロース寒天培地(TCBS)培地での分析、および特定の抗血清を用いたスライド凝集反応による同定[12]。しかし、この技術は非常に時間と労力を要し、数日後に得られた結果は、臨床診断と患者の治療の遅れを意味するでしょう。 Vの検出のためのPCR増幅を含む分子的方法。コレラ [13]は診断時間を短縮しました[14]が、PCR法は熟練した専門的で高価なインフラストラクチャを必要とし、リソースが少ない国では実行が困難です。免疫クロマトグラフィーの原理に基づく迅速な診断検査が、 Vの離散的または同時検出について報告されている。コレラ 血清型O1およびO139。 Vの検出に使用される他のいくつかのイムノアッセイベースの技術。コレラ 酵素免疫測定法(ELISA)、共凝集、免疫蛍光、水晶振動子微量天秤(QCM)などがあります。ただし、これらの手法のほとんどには、高度な機器、長いアッセイ時間、および詳細な技術知識を備えた高度な資格を持つ担当者が必要です[15、16、17、18、19、20]。
電気化学的方法は、その高感度、特異性、単純さ、および経済的なプロトコル、ならびに迅速な検出および微細加工技術との互換性のために、核酸検出においてかなりの注目を集めています[21、22]。さらに、小型化技術と組み合わせた電気化学的方法は、その場での分散分析に使用できます。たとえば、マイクロ流体チップベースのDNAバイオセンサーデバイスは、実用的な設定に非常に役立ちます[23]。ガラス状炭素電極(GCE)、カーボンペースト電極(CPE)、金電極、白金電極など、電気化学測定に使用される電極にはさまざまなものがあります。最近では、バックグラウンド電流が低く、電位窓が広い、カーボンインクが安価で大量生産できるなどの独自の特性により、スクリーンプリント電極(SPE)の使用に研究が集中しています。 。
Vの検出について報告されているいくつかの電気化学的方法があります。コレラ 一連の複雑なステップで構成されていました。電気化学的 V。コレラ Liewらによって報告されたジェノセンサー。 [24]は、電気化学的吸着法を使用して、カーボンSPEにDNAプローブを固定しました。高分子電解質を含む凍結乾燥したAuNPs修飾多層PSA粒子は、サンドイッチDNAハイブリダイゼーションアッセイでレポーターラベルとして機能するアビジンとバイオコンジュゲートを形成しました。ただし、PSA-AuNPs-アビジンバイオコンジュゲートを保存してDNAバイオセンサーの動作期間を最大30日間延長するには、ソルビトール安定剤の添加が必要でした。酵素電気化学 V。コレラ DNAバイオセンサーは最近Yuらによって考案されました。 [25]これにより、チオール化抗フルオレセイン結合アルカリホスファターゼ(抗FCAP)標識DNAプローブは、金-チオール化学によって金SPEに結合しました。標的DNAはユニバーサルフルオレセインでタグ付けされ、α-ナフチルホスフェートから電気活性α-ナフトールへの酵素的変換を介して達成されるDNAハイブリダイゼーション認識を可能にしました。それにもかかわらず、この検出スキームは、アンペロメトリー測定を行う前に、DNAハイブリダイゼーション、機能性酵素によるDNAハイブリッドの標識、続いて電気的に不活性なα-ナフチルホスフェート基質中での電極のインキュベーションに約95分の長いアッセイ時間を必要としました。ビオチン化DNAプローブと結合したアビジン被覆カーボンSPEに基づく別の酵素電気化学DNAバイオセンサーは、Lowとチームメンバーによって開発されました[26]。ジゴキシゲニン(DIG)標識レポータープローブも、cDNA配列に隣接するこのダブルハイブリダイゼーション戦略で使用されました。西洋ワサビペルオキシダーゼ結合抗DIG(抗DIG-HRP)を電気化学的標識として使用し、H の還元と同時に3,3 '、5,5'-テトラメチルベンジジン(TMB)の酸化を触媒することができました。 2 O 2 DNAハイブリダイゼーションイベントの電気化学的変換のための電極表面への電子移動をもたらす。チオール化DNAプローブで固定化された金でコーティングされたガラス電極に基づく簡単なDNAバイオセンサーの設計は、Patelらによって説明されました。 [22] Vの迅速な検出用。コレラ 、およびメチレンブルーをDNAハイブリダイゼーションインジケーターとして使用しました。ただし、システムの線形検出範囲はμMレベルに制限されているため、臨床サンプルへの適用が制限されていました。
ラテックス-金ナノ粒子は、以前は、 Vの検出におけるDNAプローブへのアビジン/ビオチン結合を介したDNAハイブリダイゼーション標識として使用されてきた。コレラ [24]、魚の病原体 Aphanomyces invadans [27]、 E。コリ [28]、および非特異的DNAハイブリダイゼーション[29]。これにより、金ナノ粒子の負に帯電したコロイドが静電的に付着する前に、ラテックス球が高分子電解質の多層でコーティングされました。 KawdeとWang [29]は、PSAラテックス粒子をDNAレポータープローブに付着させ、ストレプトアビジンでコーティングされたラテックス粒子にビオチンでコーティングされたAuNPをロードすることでDNAハイブリダイゼーションラベルとして使用しました。クアンら[24、27]およびLiew etal。 [24、27]は、金-PSA-DNAプローブコンジュゲートを使用した同じアビジン-ビオチン結合法も報告しました。 Pinijsuwan etal。 [28]は、DNAレポータープローブに付着したPSA粒子をロードするための静電法の使用を報告し、高分子電解質の金でコーティングされたPSA粒子は、DPV電流応答を増幅するハイブリダイゼーションのラベルとして使用されました。
この研究では、 Vの高感度検出システムを開発するために、DNAプローブ固定化基質としてラテックス-金ナノ粒子を使用した別のDNA固定化アプローチを報告しています。コレラ DNA。 DNAの固定化は、カルボキシル化ラテックスへの固定化効率を改善するためのカップリング試薬として1-エチル-3-(3-ジメチルアミノプロピル)カルボジイミド塩酸塩/ N-ヒドロキシスクシンイミド(EDC / NHS)化学を使用して、非常に簡単で迅速な手順で実行されました[30]粒子表面。 DNAハイブリダイゼーションの検出は、固定化されたDNAプローブとターゲット配列の間のハイブリダイゼーション反応とそれに続くシグナル/レポータープローブを含むサンドイッチタイプのアッセイに基づいていました。アントラキノン-2-スルホン酸一水和物ナトリウム塩(AQMS)は、ハイブリダイゼーションイベントを監視するための電気化学的ラベルとして使用されました。提案されたサブミクロンサイズのラテックス粒子は、DNAプローブの結合能力を改善し、DNAバイオセンサーの感度は、高導電性の金ナノ粒子(AuNP)を組み込むことで強化されました。 DNAバイオセンサーは、 Vの検出に対して並外れた感度を示した。コレラ これまでに報告されているアビジン-ビオチン技術と比較して、cDNAとゼプトモルレベルでの非常に低い検出限界[24、26]。
メソッド
化学薬品および試薬
スチレン(St)とアクリル酸(AA)はFlukaから購入しました。 HAuCl 4 ・3H 2 O、クエン酸三ナトリウム二水和物、ドデシル硫酸ナトリウム(SDS)、1-エチル-3-(3-ジメチルアミノプロピル)カルボジイミド塩酸塩(EDC)、およびN-ヒドロキシスクシンイミド(NHS)はSigma-Aldrichから入手しました。過硫酸アンモニウム(APS)、臭化水素酸、および臭素は、それぞれRiedel-DeHaën、Ajax Finechem、およびPanreacから供給されました。すべての化学溶液は脱イオン水で調製されました。 30塩基のターゲットとミスマッチの合成オリゴヌクレオチドの両方がBioService Unit(NSTDA)から調達されました。非相補的DNA(ncDNA)とシグナルプローブはSigmaからのものであり、5'-アミノ修飾キャプチャープローブはBioneerからのものでした。キャプチャープローブは0.05Mのリン酸カリウムバッファー(pH 7)で調製し、ターゲットDNA、ミスマッチターゲット、レポータープローブ、および非相補的DNA溶液はリン酸ナトリウムバッファー(0.05 M、pH 7)で調製しました。本研究で使用したオリゴヌクレオチド配列を表1に示します。
<図>装置
電気分析測定は、GPES(4.0.007)ソフトウェアを備えたポテンシオスタット/ガルバノスタット(Autolab PGSTAT12、Metrohm)を使用して実行されました。ボルタンメトリー実験は、カーボンペーストスクリーンプリント作用電極(SPE)(Quasense、バンコク、タイ)、Ag / AgCl参照電極(3 MのKCl)、およびプラチナロッド(3 MのKCl)で構成される従来の3電極システムを使用して実行されました。直径2mm)対極。微分パルスボルタンメトリー(DPV)技術は、0.02Vのステップ電位と0.5V / sのスキャンレートで、pH7および4.5mLの測定バッファー(0.05 Mのリン酸カリウムバッファー)で0.0〜 + 1.0Vのすべての電気化学的調査に使用されました。周囲温度。この研究で測定されたすべての電位はAg / AgCl電極を基準とし、超音波処理槽(Elma S30H)を使用して均質な溶液を調製しました。走査型電子顕微鏡(SEM、LEO 1450VP)を使用して、ラテックス球のサイズと分布を決定しました。
メソッド
コロイド金ナノ粒子の合成
コロイド状AuNPは、Turkevich法[31]に従ってクエン酸ナトリウムを還元することによって調製されました。簡単に説明すると、約10mLの5mM HAuCl 4 ・3H 2 Oを180mLの脱イオン水に溶解し、ホットプレートとマグネチックスターラーを組み合わせた装置で連続撹拌条件下で沸騰させました。 0.5%の10ミリリットル( w / v 次に、クエン酸三ナトリウムを沸騰溶液に加え、溶液の色が淡い赤からルビーレッドに徐々に変化するのが観察された。
ラテックス粒子の準備
ラテックス粒子は、Polpanich et al。によって説明されているように、石鹸を含まないエマルジョン共重合反応によって調製されました。 [23]いくつかの変更があります。手短に言えば、約190gの脱イオン水を、350rpmで撹拌しながら約1時間水浴に沈めた3つ口フラスコ内で窒素ガスでパージした。次に、20グラムのStと0.5 gのAAを添加し、温度を70°Cに維持しました。その後、約0.2gのAPSを10mLの脱イオン水に加え、続いて3つ口フラスコ内の配合物に注いでラジカル重合反応を開始し、重合プロセスを8時間進めた。合成されたままのカルボキシルラテックス球を、脱イオン水で2回13,000 rpmで20分間遠心分離して回収し[23、27、28]、使用するまで室温(25°C)で脱イオン水に再懸濁しました。 PSAラテックス粒子の形態と平均サイズは、走査型電子顕微鏡(SEM)によって決定されました。
SPE表面およびDNAプローブ固定化の変更
表面改質の前に、カーボンSPEを脱イオン水で完全にすすぎ、次に3 mg / mLのPSA懸濁液でドロップコートし、周囲条件で風乾させた後、5 mg / mLのコロイド状AuNPでドロップキャストしました。 。ラテックス粒子とAuNPによる修飾前後のカーボンSPEの電気化学的特性をCV法で調べた。次に、ラテックス-AuNPs修飾炭素SPE(PSA-AuNPs-SPE)を脱イオン水ですすぎ、カルボジイミド架橋試薬を含む0.1 Mリン酸カリウム緩衝液(pH 5)、つまり0.002MのEDCと0.005MのEDCに浸しました。 NHSを2時間[32]、5μMの捕捉プローブを含む0.05 Mのリン酸カリウム緩衝液(pH 7)に一晩浸します。その後、DNA修飾PSA-AuNPs-SPE(DNA-PSA-AuNPs-SPE)をリン酸カリウム緩衝液(0.05 M、pH 7)で十分に洗浄し、物理的に吸着したプローブを除去しました。 DNA電極を線形標的DNA(1μM)とAQMS DNAハイブリダイゼーションラベル(5 mM)を含むpH7の0.05Mリン酸ナトリウムバッファーに1時間浸し、その後0.05 Mのリン酸ナトリウムバッファー(pH 7)完全なDNAハイブリダイゼーションプロセスのために、1μMのレポータープローブと5mMのAQMSでさらに1時間コンディショニングします。最後に、DNA電極をリン酸カリウム緩衝液(0.05 M、pH 7)でリンスし、ハイブリダイズしていないオリゴヌクレオチド配列を除去しました。 SPEに付着した各伸長物質の電気化学的応答をDPV法で調べた。図1は、 Vを開発するための段階的な手順を表しています。コレラ コロイド状PSA-AuNPs固体支持体に基づくDNAバイオセンサー。
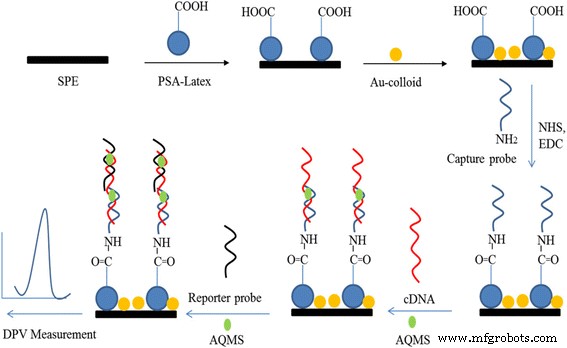
DNAバイオセンサーの段階的製造手順の概略図
合成オリゴヌクレオチドハイブリダイゼーションの最適化
DNAの動的線形範囲を決定する前に、固定化されたDNAプローブとシグナルプローブおよびターゲットDNAとのハイブリダイズ応答に対する、DNAプローブおよびAQMS濃度、pH、イオン強度、バッファー容量などのさまざまなパラメーターの影響を調べました。バイオセンサー。さらに、捕捉プローブの固定化およびDNAハイブリダイゼーションの持続時間、ならびにバイオセンサーの寿命および再生もまた、開発されたVの前に評価された。コレラ DNAバイオセンサーは、スパイクアンドリカバリー実験への応用の準備ができていました。キャプチャープローブとAQMSのローディングは、それぞれ1〜6μMと0.1〜5 mMの濃度に変化させることで最適化され、ターゲットDNAとレポータープローブの濃度は0.05 Mリン酸ナトリウム緩衝液(pH 7.0)で5μMに維持されました。リン酸ナトリウム緩衝液のpHと濃度をそれぞれpH5.5から8.0および0.001から1.000Mに変更することにより、pH効果と緩衝液濃度の研究を実施しました。電気化学的DNAバイオセンサーのDNAハイブリダイゼーション応答における異なるカチオンの存在は、Na + を加えることによって実行されました。 、K + 、Ca 2+ 、およびFe 3+ 1mMのAQMSと5μMのcDNAおよびpH7.0の検出プローブを含むDNAハイブリダイゼーションバッファーへの1.0Mのイオン。イオン強度の影響は、pH 7.0で0.1〜3.0Mの範囲でNaCl濃度を変化させることによって調べられました。次に、DNAバイオセンサーのダイナミックレンジを1.0×10 -21 で決定しました。 〜1.0×10 -8 M V。コレラ 5μMおよびpH7.0で一定のシグナルプローブ濃度を使用するcDNA。 DNAプローブの固定化時間は、DNA電極を5μMのキャプチャープローブ溶液(pH 7.0)に1〜13時間浸すことによって決定され、DPV応答は1〜2時間ごとに取得されました。一方、DNAハイブリダイゼーション時間は15〜180分の間にDNAハイブリダイゼーション反応が起こることを可能にすることによって決定され、DNAバイオセンサー応答は15〜30分ごとに記録されました。 DNAバイオセンサーの貯蔵寿命は、5μM Vの検出に向けたDNAバイオセンサー応答を定期的に測定することによって決定された。コレラ 120日間のcDNA。分析は、サンドイッチハイブリダイゼーションアッセイごとに新しいDNA電極を使用して5回繰り返して実施しました。 DNA電極の再生は0.1MのNaOH溶液を使用して4分間行い、DNAバイオセンサーの再ハイブリダイゼーション(60分)は5μMのcDNAと検出プローブおよび1mMのAQMSを含む2.0Mイオン強度の再ハイブリダイゼーション溶液を使用して行いました。 0.05Mのリン酸カリウム緩衝液(pH7.0)中。再生実験は6回繰り返して行われました。
V。コレラ PSA-AuNPsベースの電気化学的DNAバイオセンサーを使用した定量化
さまざまな V。コレラ 細菌株、すなわちJ2126-I、J2126-II、J3324-I、J3324-II、J3330-I、J3330-II、CDHI 5294-II、および Citroobacter freundii を含むUVC1324 (CF-I)および Citroobacter freundii (CF-II)は、クダ州のAIMST大学応用科学部の微生物学研究所から入手しました。次に、QIAGEN Genomic-tip 500 / Gを製造元のプロトコルに従って使用して、これらの細菌に対してゲノムDNA抽出を行いました。次に、抽出したDNAをリン酸ナトリウム緩衝液(0.05 M、pH 7.0)を使用して100倍に希釈しました。 2.0MのNaClと1mMのAQMSを含む約300mLの抽出されたDNAを15分間超音波処理して、DNA切断を解放しました。次に、固定化されたDNAプローブを1時間浸して、DNAハイブリダイゼーションプロセスを実行し、0.05 Mリン酸カリウムバッファー(pH 7.0)で注意深く洗浄して、結合していないDNAを除去しました。 DPVピーク電流に基づくDNAバイオセンサー応答の評価を測定し、cDNAを制御信号として反応させずにDNA電極によって生成された電流応答と比較しました。各実験は、同じ実験条件下で三重に実施された。一般的な t テストは、DNAバイオセンサーの応答とコントロールの応答の間の有意差を決定するために使用されました。 Vの回復。コレラ J3324および V。コレラ 1.0×10 -4 のUVC1324DNA μgμL -1 、1.0×10 -5 μgμL -1 、および1.0×10 -6 μgμL -1 次に、ハイブリダイゼーションバッファーにスパイクしたものを、提案されたPSA-AuNPsベースの電気化学DNAバイオセンサーを使用して実行しました。
結果と考察
ラテックス粒子の形態とラテックスゴールドナノ粒子修飾SPEの電気化学的挙動。
図2は、平均粒子サイズが186.1±4.6nmのカルボキシル化ラテックス球のSEM顕微鏡写真を示しています。 PSA球の均一なサイズ分布により、ラテックス表面へのDNA分子の均一な固定化が可能になり、DNAバイオセンサーの再現性応答が向上しました。走査型電子顕微鏡(SEM、LEO 1450VP)を使用して、ラテックス球のサイズと分布を決定しました。

10,000倍の倍率で合成されたままのPSAラテックス球のSEM顕微鏡写真
変更されたSPEの電気力学的結果を表2に示します。ピーク電位分離(ΔEp)は、システムの電子移動速度を示します。 PSA修飾SPE(PSA-SPE)は、コロイド共重合体粒子層での電荷移動プロセスが遅いために最も高いΔEp値を示し、システムを準可逆状態に移行させました。ただし、AuNPがPSA-SPEにロードされた場合、ΔEpの減少は、電極表面での電子移動速度の向上を意味します。
<図>図3は、ラテックス修飾SPEでのAQMS酸化応答の微分パルスボルタモグラムと Vのシーケンシャルハイブリダイゼーション応答を示しています。コレラ DNAバイオセンサー。ラテックス修飾ミクロスフェアのみを含み、固定化された捕捉DNAを含まない電極(実験(a)および(b))と、cDNAおよび報告されたプローブの存在下で固定化された捕捉DNAプローブで修飾された電極(実験(g ))。これは、アミノ化されたDNAキャプチャープローブが、EDC / NHSカップリングプロトコルを介してコーティングされたカルボキシル化PSAラテックス球にうまく固定化されたことを示しています[33]。実験(g)は、ncDNAと報告されたレポータープローブ(実験(d))の存在下、ミスマッチDNAと報告されたレポータープローブ(実験(e))、およびターゲットcDNAの存在下で、DNA電極と比較してはるかに高いDPV応答も示しています。プローブは報告されていません(実験(f))。これは、実験(g)で示されているように、DNAバイオセンサー表面でのサンドイッチハイブリダイゼーション反応による、ターゲットDNAとキャプチャーおよびレポータープローブの完全なハイブリダイゼーションによるものです。これはまた、報告されたプローブの使用がDNAハイブリダイゼーションからのシグナルを増強する可能性があることを示しています。それにもかかわらず、報告されたプローブを組み込まずに標的DNAの存在下で観察されたハイブリダイゼーションから生じたDPV電流(実験(f))は、ハイブリダイズしていないDNAで観察されたDPV電流シグナル(実験(c)、(d)、および(e))。
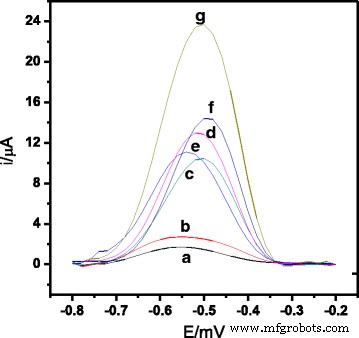
電極のAQMSからの差動パルスボルタモグラム信号( a )PSA-SPE、( b )PSA-AuNPs-SPE、( c )プローブ-PSA-AuNPs-SPEをキャプチャし、( d )ncDNAおよびレポータープローブ、( e )ミスマッチDNAとレポータープローブ、( f )cDNAのみ、および( g )cDNAおよびレポータープローブ
DNAプローブのローディングとAQMS濃度の影響
DNAハイブリダイゼーション応答に対するDNAプローブ濃度の影響は、AQMS電気化学的酸化応答を通じて観察されました。図4aは、PSA-AuNPs-SPEに固定化されたDNAプローブ量が1から4μMに増加するにつれて、DNAバイオセンサー応答が徐々に増加したことを示しています。これは、固定化されたDNAヘリックスを介した電子移動を行うために二本鎖DNA(dsDNA)に挿入された電気活性AQMSの量の増加に起因していました。 DNAバイオセンサーのDPV応答は、4〜6μMのDNAプローブ間でほぼプラトーになることが観察されました。これは、電極表面への最適なDNAプローブのローディングが達成されたことを示しています[34]。したがって、4μMのキャプチャープローブが、後続の実験で最適なDNAプローブローディングとして選択されました。 AQMSラベルの濃度も、0.1〜5.0 mMの測定電解質で最適化されており、1 mMのAQMSの濃度は、最適なDNAインターカレーション反応に十分であることがわかりました(図4b)。
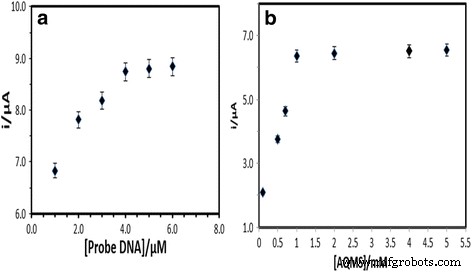
キャプチャープローブの効果( a )およびAQMS濃度( b )0.05 Mリン酸ナトリウム緩衝液(pH 7.0)中の5μMcDNAおよびシグナルプローブで実行されたDNAバイオセンサー応答について
pH、イオン強度、およびバッファー容量の影響
DNAハイブリダイゼーション反応の速度は、溶液のpHに大きく依存します。図5aに見られるように、より酸性の環境下では、DNAのホスホジエター骨格のプロトン化によりDNA分子の溶解度が低下し、最終的にDNAハイブリダイゼーション反応速度が低下しました。一方、塩基性媒体では、DNA塩基対を一緒に保持している弱い水素結合を切断しました。最適なDNAハイブリダイゼーション反応は中性条件でより有利であり、それにより、より多くのキャプチャープローブが標的DNAとハイブリダイズするように促進され、その後、AQMSレドックスプローブのインターカレーションが可能になり、DNAハイブリダイゼーション認識がビジネスになりました。したがって、pH7.0の0.05Mのリン酸ナトリウム緩衝液を、その後のDNAバイオセンサー研究のためのDNAハイブリダイゼーション培地として使用した。 Ca 2+ などの正に帯電したイオン 、Na + 、K + 、およびFe 3+ イオンは、負に帯電したDNAのホスホジエステル鎖と相互作用する可能性があります。このイオン反応はDNA分子の電荷を中和し、DNA分子間の立体反発を減らしてDNAハイブリダイゼーション反応を促進します。図5bは、DNAハイブリダイゼーション応答に対するいくつかのカチオンの影響を示しています。 DNAハイブリダイゼーション応答は、Na + の順序で正に帯電したイオンの存在下で増加することに気づきました。> K + > Fe 3+ > Ca 2+ 。両方のCa 2+ およびFe 3+ Ca 2+ のイオン相互作用により、イオンはDNAハイブリダイゼーション応答を大幅に低下させることがわかりました。 およびFe 3+ 緩衝液からのリン酸イオンを含むイオン。これにより、不溶性のリン酸化合物が形成されます。これにより、培地のイオン含有量が減少し、それによってDNA分子間の静電反発力が増加しました。最高のDNAハイブリダイゼーション電流はNa + の存在下で得られました K + と比較して、サイズが小さく、DNA糖リン酸骨格に対する親和性が強いため DNAの負に帯電したリン酸基間の立体障害と静電反発を克服するためのイオン。
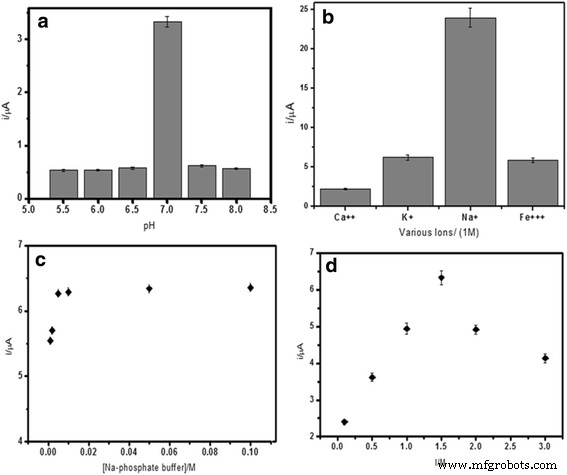
pHの影響( a )さまざまな陽イオン( b )、バッファー濃度( c )、およびイオン強度( d )電気化学的 VのDNAハイブリダイゼーション応答について。コレラ DNAバイオセンサー。ハイブリダイゼーションは5μMのcDNAとレポータープローブを用いて行い、続いて1mMAQMSを用いてインターカレーションを行いました
さらに、溶液のイオン強度もDNAバイオセンサーの応答に影響を与えます。図5c、dは最適なバッファー容量を示し、イオン強度は0.05Mのリン酸ナトリウムバッファーを使用して達成されました。pHはそれぞれpH7.0および2.0MのNaClに固定されています。この状態では、DNAハイブリダイゼーション反応が最も良好になりました。したがって、高いDPV応答が得られました。溶液の最適な緩衝能とイオン強度で、DNA分子間の静電反発力が減少し、DNAハイブリダイゼーション反応が改善されました。対照的に、低すぎるまたは高すぎるイオン含有量を使用すると、立体障害と静電反発力が支配的になり、DNA分子の混成が制限されました。
Vの確立。コレラ DNAバイオセンサーの検量線
図6aに示す結果から、DNAバイオセンサーの応答は、cDNA濃度が1.0×10 -21 から増加するのに比例して増加しました。 〜1.0×10 -8 M( R 2 =0.99)検出限界1.0×10 −21 M.検出限界は、検出限界を線形較正勾配で割ったものに近似する応答曲線でのバイオセンサー応答の標準偏差の3倍に基づいて計算されました。 DNAバイオセンサーの広い線形検出範囲は、DNA固定化のキャリアマトリックスとして使用されるサブミクロンサイズの範囲の高度に単分散で球状のPSAラテックス粒子によるものでした。ラテックス粒子表面のアクリル酸に富む層は、DNA捕捉プローブを付着させるための大きな結合部位を提供し、DNA受容層によって最大の被覆表面を作成しました。さらに、PSA修飾SPEにAuNPを組み込むと、DNAハイブリダイゼーション応答の分析シグナルがさらに増幅され、DNAバイオセンサーの感度が高くなりました(図6b)。
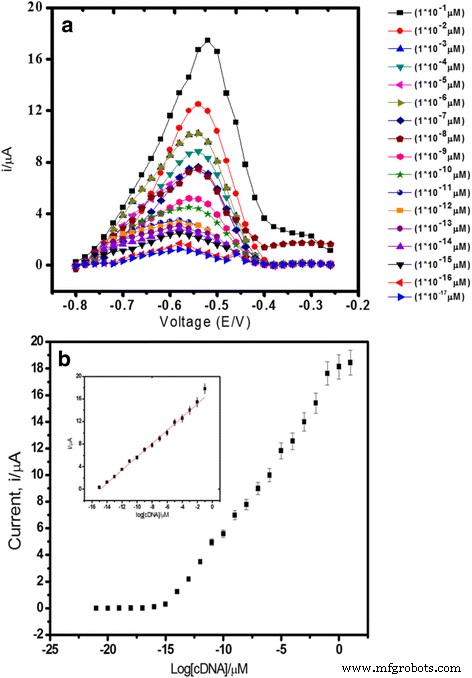
Differential pulse voltammograms (a ) and DNA biosensor linear range (b ) obtained using various cDNA concentrations from 1.0 × 10 −15 to 1.0 × 10 −1 μM V. cholerae target DNA and 5 μM signal probe
DNA Probe Immobilisation and Hybridization Times
It took about 8 h for the capture probe to be immobilised on the PSA copolymer particles surface, as illustrated by the DNA biosensor response in Fig. 7a, which showed a current increment from 1.0 to 8.0 h of capture probe immobilisation time, after which no obvious change in the DPV current was observed. Longer immobilisation time resulted in a higher amount of DNA probes immobilised onto the latex. After 8.0 h of exposure to the DNA probes, the hydrophilic functional latex with reactive carboxyl groups at the surface was presumably fully attached with the DNA probes. DNA hybridisation time, on the other hand, is the rate limiting step, which determines the response time of the DNA biosensor. Based on the DNA biosensor response trend in Fig. 7b, the response time of the V. cholerae DNA biosensor developed in this study was estimated to be about 60 min for the dual hybridisation processes to complete.
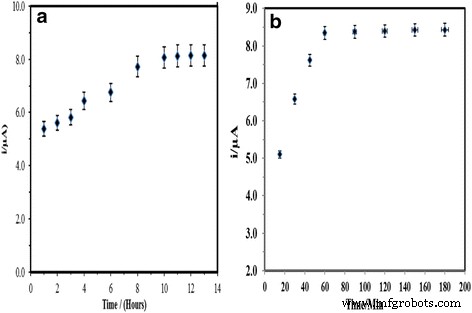
DNA probe immobilisation duration on the immobilised PSA latex colloidal particles (a ) and DNA hybridization duration of the DNA biosensor (b ) in 0.05 M potassium phosphate buffer at pH 7.0 containing 5 μM target DNA and reporter probe and 1 mM AQMS at 2.0 M ionic strength
Long-Term Stability and Regeneration of V.cholerae DNA Biosensor
Figure 8 shows the shelf life of the V. cholerae DNA biosensor. The DNA biosensor showed the highest response to the detection of 5 μM of V. cholerae cDNA for the first month of the experimental period. The electrochemical DNA biosensor was able to retain 95% of its initial DPV current after 58 days of storage period. The DNA hybridisation response was then gradually decreased to about 75% of its original response on the 75th day and exhibited 40% of its initial performance on the 100th operational day. The bioactivity of the immobilised capture probe was finally declined to 30% after 3 months of storage period. The reproducibility of each calibration point, which was repeated on five replicate DNA electrodes, gave satisfactory relative standard deviation (RSD) between 2.4 and 4.5% (n =5).
The life span profile of the fabricated V. cholerae DNA sensing electrode. The electrode was stored in 0.05 M potassium phosphate buffer (pH 7.0) at 4 °C after every DPV measurement was taken
Regeneration of biosensor indicates whether the biosensor is reusable for a series of consecutive analyses. The regeneration method used in this study was conducted based on previously reported protocol in other studies [11, 21] with slight modifications. In this study, 0.1 M of NaOH solution was used as the regeneration solution to break the hydrogen bonds between base pairs of hybridised dsDNA. With the result from Fig. 9, it is notable that the DNA biosensor response declined significantly after incubation in 0.1 M of NaOH and the percentage of the DNA biosensor response reduced from 35.1 to 5.2% relative to the DNA biosensor initial response after incubation in 0.1 M of NaOH solution from 30 to 240 s. The DNA biosensor response decreased with the increasing incubation time signifies the hydrogen bonds between hybridised dsDNA were broken up by the alkaline regeneration solution. However, rehybridisation of the DNA biosensor was able to attain almost 100% of its initial response for a consecutive six DNA analyses with a reversibility RSD of 5%.
Repeatability of V. cholerae DNA biosensor using 0.1 M NaOH regeneration solution and rehybridization solution containing 5 μM cDNA and detection probe and 1 mM AQMS at 2.0 M ionic strength in 0.05 M potassium phosphate buffer (pH 7.0)
Determination of V. cholerae Bacteria with the Developed DNA Biosensor
The optimised DNA biosensor has been applied to quantify the V. cholerae DNA extracted from various V. cholerae bacterial strains. Table 3 presents the results acquired from DNA tests carried out with the hybridisation medium spiked with different strains of V. cholerae DNAs and other bacterial species at a concentration within the calibration range of the DNA biosensor. The DNA biosensor showed superior selectivity towards V. cholerae J3324–I, V. cholerae J3324–II, and V. cholerae UVC1324 with high DPV current response and low current signals were obtained for the evaluation of both Citrobacter freundii (CF-I) and Citrobacter freundii (CF-II).
<図>Recovery of V. cholerae J3324 and V. cholerae UVC1324 DNAs at three different concentrations spiked into the hybridisation buffer demonstrated 91.4 ± 2.2% to 108.9 ± 4.8% (n =3) of recoveries percentage (Table 4). This result suggests that the proposed PSA-AuNPs-based electrochemical DNA biosensor could be adopted for highly reliable and accurate detection of V. cholerae DNA in environmental and clinical samples.
<図>Performance Comparison with Other Reported V. cholerae DNA Biosensors
Based on the data summarised in Table 5, the proposed electrochemical DNA biosensor based on PSA-AuNPs immobilisation material shows an exceptional broad linear quantification range compared to other planar two-dimensional electrodes as the DNA supporters. This clearly demonstrates the advantage of the micro-sized latex particles where the polymeric PSA is capable to intensify the probe binding capacity with a simple loading method via the classical EDC/NHS coupling compared to avidin-biotin technology [24, 26] and ultra-low detection limit in zeptomolar range with reasonable assay time.
<図>Conclusions
This study reports the development of an electrochemical DNA biosensor for the detection of one of the most devastating high-risk V. cholerae pathogens. The PSA-AuNPs-modified DNA biosensor can be used for direct detection of DNA of interest from the extracted DNA without the need of amplification reaction via conventional PCR method, which is commonly used in those previously reported V. cholerae DNA biosensors. In addition, no further dilution of the extracted DNA is needed as the high-capacity AuNPs-doped latex microspheres-based DNA biosensor is highly sensitive for the quantitation of DNA at extremely low level in sub zeptomolar range. Therefore, the electrochemical DNA biosensor is greatly suitable as a surveillance and diagnostic tool to control the epidemic of the fatal intestinal infection.
ナノマテリアル
- ビジュアルデータをIoTと統合する可能性
- 卵巣癌細胞の高感度かつ迅速な検出のための柔軟なグラフェンベースのバイオセンサーのデモンストレーション
- 金ナノ粒子修飾ガラス状炭素電極上の尿酸の強化された光電気化学的検出
- アロワナの魚の性別を測定するためのアクリルゴールドナノコンポジットからの高感度電気化学DNAバイオセンサー
- スーパーキャパシターの電極材料としての階層的多孔質構造を持つ単分散カーボンナノスフェア
- ポリ(4-スチレンスルホン酸-co-マレイン酸)による金磁性ナノ粒子の安定性の向上:タンパク質検出用に調整された光学特性
- アップコンバージョン発光を増強するための異なる形態のBaYF5:Er3 +、Yb3 +の制御された合成
- CCRF-CEMのターンオン検出のための酸化グラフェンベースの蛍光アプタセンサー
- 柔軟なひずみセンサー用の高い伸縮性を備えた導電性TPUナノファイバー複合材料
- 高速で室温の直接テラヘルツ検出のための大面積で均一なグラフェンナノメッシュの製造
- Impossible Objectsは、複合3D印刷のためにBASFと提携しています



